这篇文章主要介绍了R语言中生存分析模型与时间依赖性ROC曲线可视化,本文给大家介绍的非常详细,对大家的学习或工作具有一定的参考借鉴价值,需要的朋友可以参考下
R语言简介
R是用于统计分析、绘图的语言和操作环境。R是属于GNU系统的一个自由、免费、源代码开放的软件,它是一个用于统计计算和统计制图的优秀工具。
人们通常使用接收者操作特征曲线(ROC)进行二元结果逻辑回归。但是,流行病学研究中感兴趣的结果通常是事件发生时间。使用随时间变化的时间依赖性ROC可以更全面地描述这种情况下的预测模型。
时间依赖性ROC定义
令 Mi为用于死亡率预测的基线(时间0)标量标记。 当随时间推移观察到结果时,其预测性能取决于评估时间 t。直观地说,在零时间测量的标记值应该变得不那么相关。因此,ROC测得的预测性能(区分)是时间t的函数 。
累积病例
累积病例/动态ROC定义了在时间t 处的阈值c处的 灵敏度和特异性, 如下所示。
累积灵敏度将在时间t之前死亡的视为分母(疾病),而将标记值高于 c 的作为真实阳性(疾病阳性)。动态特异性将在时间t仍然活着作为分母(健康),并将标记值小于或等于 c 的那些作为真实阴性(健康中的阴性)。将阈值 c 从最小值更改为最大值会在时间t处显示整个ROC曲线 。
新发病例
新发病例ROC1在时间t 处以阈值 c定义灵敏度和特异性, 如下所示。
累积灵敏度将在时间t处死亡的人 视为分母(疾病),而将标记值高于 Ç 的人视为真实阳性(疾病阳性)。
数据准备
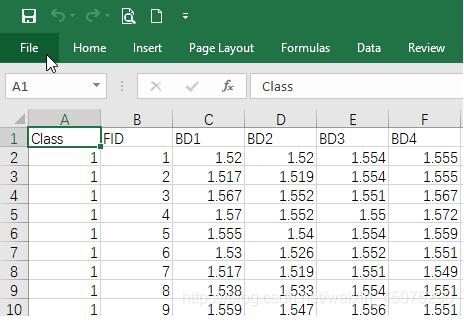
我们以数据 包中的dataset3survival为例。事件发生的时间就是死亡的时间。Kaplan-Meier图如下。
## 变成data_frame
data <- as_data_frame(data)
## 绘图
plot(survfit(Surv(futime, fustat) ~ 1,
data = data)可视化结果:
在数据集中超过720天没有发生任何事件。
## 拟合cox模型
coxph(formula = Surv(futime, fustat) ~ pspline(age, df = 4) +
##获得线性预测值
predict(coxph1, type = "lp")累积病例
实现了累积病例
## 定义一个辅助函数,以在不同的时间进行评估
ROC_hlp <- function(t) {
survivalROC(Stime
status
marker
predict.time = t,
method = "NNE",
span = 0.25 * nrow(ovarian)^(-0.20))
}
## 每180天评估一次
ROC_data <- data_frame(t = 180 * c(1,2,3,4,5,6)) %>%
mutate(survivalROC = map(t, survivalROC_helper),
## 提取AUC
auc = map_dbl(survivalROC, magrittr::extract2, "AUC"),
## 在data_frame中放相关的值
df_survivalROC = map(survivalROC, function(obj) {
## 绘图
ggplot(mapping = aes(x = FP, y = TP)) +
geom_point() +
geom_line() +
facet_wrap( ~ t) +可视化结果:
180天的ROC看起来是最好的。因为到此刻为止几乎没有事件。在最后观察到的事件(t≥720)之后,AUC稳定在0.856。这种表现并没有衰退,因为高风险分数的人死了。
新发病例
实现新发病例
## 定义一个辅助函数,以在不同的时间进行评估
## 每180天评估一次
## 提取AUC
auc = map_dbl(risksetROC, magrittr::extract2, "AUC"),
## 在data_frame中放相关的值
df_risksetROC = map(risksetROC, function(obj) {
## 标记栏
marker <- c(-Inf, obj[["marker"]], Inf)
## 绘图
ggplot(mapping = aes(x = FP, y = TP)) +
geom_point() +
geom_line() +
geom_label(data = risksetROC_data %>% dplyr::select(t,auc) %>% unique,
facet_wrap( ~ t) +可视化结果:
这种差异在后期更为明显。最值得注意的是,只有在每个时间点处于风险集中的个体才能提供数据。所以数据点少了。表现的衰退更为明显,也许是因为在那些存活时间足够长的人中,时间零点的风险分没有那么重要。一旦没有事件,ROC基本上就会趋于平缓。
结论
总之,我们研究了时间依赖的ROC及其R实现。累积病例ROC可能与风险 (累积发生率)预测模型的概念更兼容 。新发病例ROC可用于检查时间零标记在预测后续事件时的相关性。
参考
Heagerty,Patrick J. and Zheng,Yingye, Survival Model Predictive Accuracy and ROC Curves,Biometrics,61(1),92-105(2005). doi:10.1111 / j.0006-341X.2005.030814.x.
到此这篇关于详解R语言中生存分析模型与时间依赖性ROC曲线可视化的文章就介绍到这了,更多相关R语言生存分析曲线可视化内容请搜索编程学习网以前的文章希望大家以后多多支持编程学习网!
本文标题为:详解R语言中生存分析模型与时间依赖性ROC曲线可视化


- Go Web开发进阶实战(gin框架) 2023-09-06
- R语言-如何切换科学计数法和更换小数点位数 2022-11-23
- Golang http.Client设置超时 2023-09-05
- 汇编语言程序设计之根据输入改变屏幕颜色的代码 2023-07-06
- Ruby的字符串与数组求最大值的相关问题讨论 2023-07-22
- Ruby 迭代器知识汇总 2023-07-23
- Ruby on Rails在Ping ++ 平台实现支付 2023-07-22
- Swift超详细讲解指针 2023-07-08
- R语言绘图数据可视化pie chart饼图 2022-12-10
- R语言关于二项分布知识点总结 2022-11-30